PCA vs t-SNE vs UMAP:可视化数据中的不可见部分
想象一下,试图通过微型望远镜探索星系——这就是机器学习模型处理原始高维数据时的感受。,其中的轴(主成分)使方差最大化。这就像旋转数据集,使其与能够捕捉最大扩散的方向对齐。💡 是的,我们将用一份备忘单、比较视觉效果和精心挑选的资源来加深您的理解。“降维就像给机器装上眼睛——它最终看到的是森林,而不仅仅是树木。它就像 t-SNE 的更智能、更快速的“表亲”。t-SNE 将高维数据映射到二维或三维空间
·
“降维就像给机器装上眼睛——它最终看到的是森林,而不仅仅是树木。”
为什么降维很重要
想象一下,试图通过微型望远镜探索星系——这就是机器学习模型处理原始高维数据时的感受。降维将复杂的高维数据集转换为更简单的形式,帮助人类和机器发现隐藏的模式。
但是有这么多可用的技术,您应该使用哪一种呢?
在此博客中,我们将从以下方面比较 PCA、t-SNE 和 UMAP :
- 直觉
- 工作原理
- 真实用例
- 视觉示例
- Python 实现
- 优点、缺点和最佳方案
💡 是的,我们将用一份备忘单、比较视觉效果和精心挑选的资源来加深您的理解。
1.主成分分析(PCA)
直觉
PCA将数据投影到一个新的坐标系中,其中的轴(主成分)使方差最大化。这就像旋转数据集,使其与能够捕捉最大扩散的方向对齐。
当您需要速度、可解释性以及与 ML 模型的兼容性时,请使用 PCA 。
PCA 的工作原理
- 标准化数据
- 计算协方差矩阵
- 查找特征向量和特征值
- 将数据投影到 top-k 个组件上
真实用例
- 金融:风险建模、信用评分
- 基因组学:减少基因表达特征
- 预处理:聚类或建模之前
Python代码
from sklearn.decomposition import PCA
from sklearn.preprocessing import StandardScaler
from sklearn.datasets import load_iris
import seaborn as sns
import matplotlib.pyplot as plt
iris = load_iris()
X = StandardScaler().fit_transform(iris.data)
y = iris.target
pca = PCA(n_components=2)
X_pca = pca.fit_transform(X)
sns.scatterplot(x=X_pca[:, 0], y=X_pca[:, 1], hue=iris.target_names[y])
plt.title("PCA on Iris Dataset")
plt.show()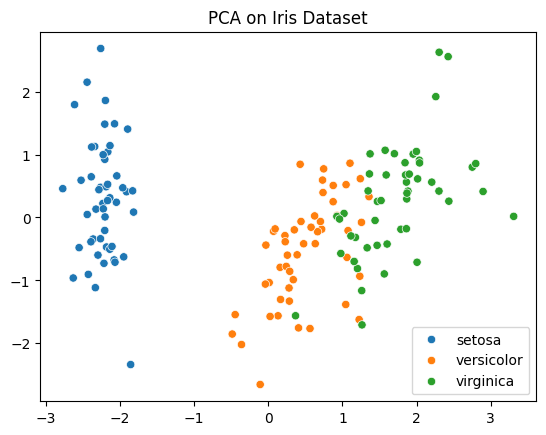
优点
- 非常快
- 保留全局结构
- 机器学习友好型
缺点
- 仅线性
- 没有聚类洞察
2. t-SNE(t分布随机邻域嵌入)
直觉
t-SNE 将高维数据映射到二维或三维空间,保留局部邻域- 这意味着相似的点保持接近。
使用 t-SNE 进行聚类可视化和探索性分析。
t-SNE 的工作原理
- 计算高维空间中的相似性
- 使用 Student-t 分布对低维相似性进行建模
- 尽量减少两者之间的分歧
真实用例
- 计算机视觉:可视化 CNN 嵌入
- 生物信息学:细胞类型聚类
- 分析:应用行为细分
Python代码
from sklearn.manifold import TSNE
tsne = TSNE(n_components=2, random_state=42, perplexity=30, n_iter=300)
X_tsne = tsne.fit_transform(X)
sns.scatterplot(x=X_tsne[:, 0], y=X_tsne[:, 1], hue=iris.target_names[y])
plt.title("t-SNE on Iris Dataset")
plt.show()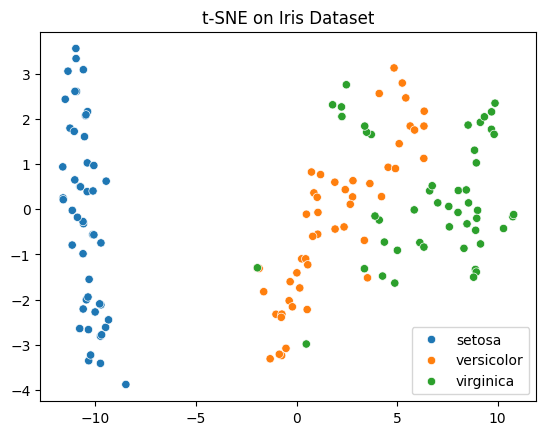
优点
- 非常适合可视化集群
- 很好地捕捉局部结构
缺点
- 计算成本高昂
- 无逆变换或机器学习兼容性
- 对超参数敏感
3. UMAP(均匀流形近似和投影)
直觉
UMAP 利用图论和流形学习,保留局部和部分全局结构。它就像 t-SNE 的更智能、更快速的“表亲”。
当您需要速度 + 集群可视化 + 可扩展性时,请使用 UMAP 。
UMAP 的工作原理
- 在高维空间中构建模糊图
- 优化低暗空间的布局
- 以最小的扭曲保留关系
真实用例
- NLP:词嵌入可视化
- 医学成像:特征提取
- 营销:客户细分
更多推荐
 已为社区贡献4条内容
已为社区贡献4条内容







所有评论(0)