代码开源!MEBOCOST:scRNA-seq + mCCC 推断 + 可视化整合实战
MEBOCOST是一种用于从单细胞RNA测序(scRNA-seq)数据中定量推断代谢物介导的细胞间通讯的算法。该方法识别代谢物(如脂质)由发送细胞合成并分泌,随后作用于接收细胞传感器蛋白(包括细胞表面受体跨膜转运蛋白与核受体)的过程。依托一个人工整理的代谢物-传感器配对数据库,MEBOCOST 根据合成酶与传感器表达量,系统判定细胞间的代谢信号传递关系。在小鼠棕色脂肪组织(BAT)中,MEBOCO
单细胞RNA测序(scRNA-seq) 技术推动了对 细胞异质性 与 组织微环境 的深入探索,但现有通讯推断方法主要集中于 配体-受体对,忽视了 代谢物介导的信号通路。MEBOCOST 引入一种整合 scRNA-seq 数据 与 代谢物-传感器数据库 的策略,系统建模 代谢物从合成到感应 的跨细胞传递过程,识别潜在的 代谢通讯网络。

- 标题:MEBOCOST:基于单细胞转录组的代谢物介导的细胞通讯建模
- DOI:https://doi.org/10.21203/rs.3.rs-2092898/v1
这个目前只是预印本,不过工具开源了,且这种算法大家可以学习一下!!
摘要
MEBOCOST 是一种用于从 单细胞RNA测序(scRNA-seq)数据 中定量推断 代谢物介导的细胞间通讯 的算法。该方法识别代谢物(如 脂质)由 发送细胞合成并分泌,随后作用于 接收细胞传感器蛋白(包括细胞表面受体、跨膜转运蛋白与核受体)的过程。依托一个人工整理的代谢物-传感器配对数据库,MEBOCOST 根据 合成酶与传感器表达量,系统判定细胞间的代谢信号传递关系。
在小鼠棕色脂肪组织(BAT)中,MEBOCOST 不仅复现了已知的代谢通讯,还发现了一批受冷刺激调控的新型通讯模式,为研究多种生理及疾病模型下的代谢通讯网络提供了重要工具。
📦 开源地址:https://github.com/zhengrongbin/MEBOCOST
结果
数据准备与 MEBOCOST 对象创建
MEBOCOST 支持两种方式导入表达数据:使用 Scanpy adata 对象(推荐)或 直接读取外部文件(如 CSV)。在此过程中,需要指定细胞类型注释、代谢估计方法、配置文件路径等参数。
## 使用 adata 对象创建:
import scanpy as sc
from mebocost import mebocost
adata = sc.read_h5ad('data/demo/raw_scRNA/demo_HNSC_200cell.h5ad')
mebo_obj = mebocost.create_obj(
adata=adata,
group_col=['celltype'],
met_est='mebocost',
config_path='./mebocost.conf',
species='human',
sensor_type=['Receptor', 'Transporter', 'Nuclear Receptor'],
cutoff_exp='auto',
cutoff_met='auto',
cutoff_prop=0.25,
thread=8
)
## 使用外部文件创建:
import pandas as pd
from mebocost import mebocost
exp_mat = pd.read_csv('your_exp_data_file.csv')
cell_ann = pd.read_csv('your_cell_ann_data_file.csv')
mebo_obj = mebocost.create_obj(
adata=None,
exp_mat=exp_mat,
cell_ann=cell_ann,
group_col=['celltype'],
met_est='mebocost',
config_path='./mebocost.conf',
species='human',
sensor_type=['Receptor', 'Transporter', 'Nuclear Receptor'],
cutoff_exp='auto',
cutoff_met='auto',
cutoff_prop=0.25,
thread=8
)
推断代谢物介导的细胞通讯(mCCC)
## 先计算代谢物的酶表达
mebo_obj._load_config_()
mebo_obj.estimator()
## 执行代谢通讯推断
commu_res = mebo_obj.infer_commu(
n_shuffle=1000,
seed=12345,
pval_method='permutation_test_fdr',
pval_cutoff=0.05
)
整合 COMPASS 进行代谢流约束分析(可选)
updated_res = mebo_obj._ConstainFlux_(
compass_folder='data/demo/compass_res/',
efflux_cut='auto',
influx_cut='auto',
inplace=False
)
mebo_obj.commu_res = updated_res
校正血液代谢物浓度影响(可选)
corrected_commu_res = mebo_obj._blood_correct_test_(
met_cont_file='data/mebocost_db/common/hmdb_blood_metabolite_concentration.tsv',
commu_score_col='Commu_Score'
)
mebo_obj.commu_res = corrected_commu_res.copy()
可视化通讯网络
通讯事件柱状图(Sender/Receiver 对数统计)
mebo_obj.eventnum_bar(
sender_focus=[],
metabolite_focus=[],
sensor_focus=[],
receiver_focus=[],
xorder=[],
and_or='and',
pval_method='permutation_test_fdr',
pval_cutoff=0.05,
comm_score_col='Commu_Score',
comm_score_cutoff=0,
cutoff_prop=0.25,
figsize='auto',
save='mebocost_eventnum.pdf',
show_plot=True,
show_num=True,
include=['sender-receiver'],
group_by_cell=True,
colorcmap='tab20',
return_fig=False
)
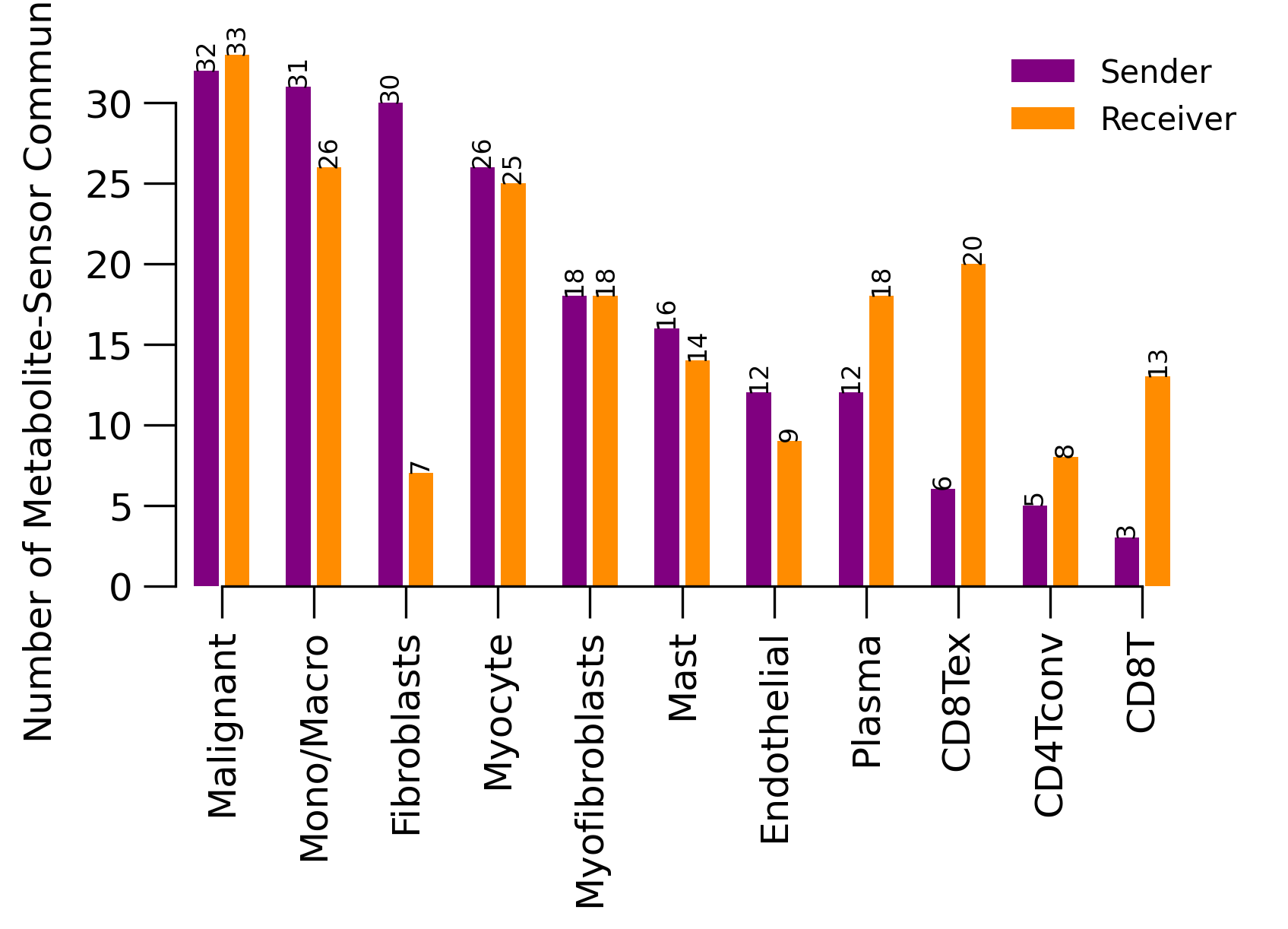
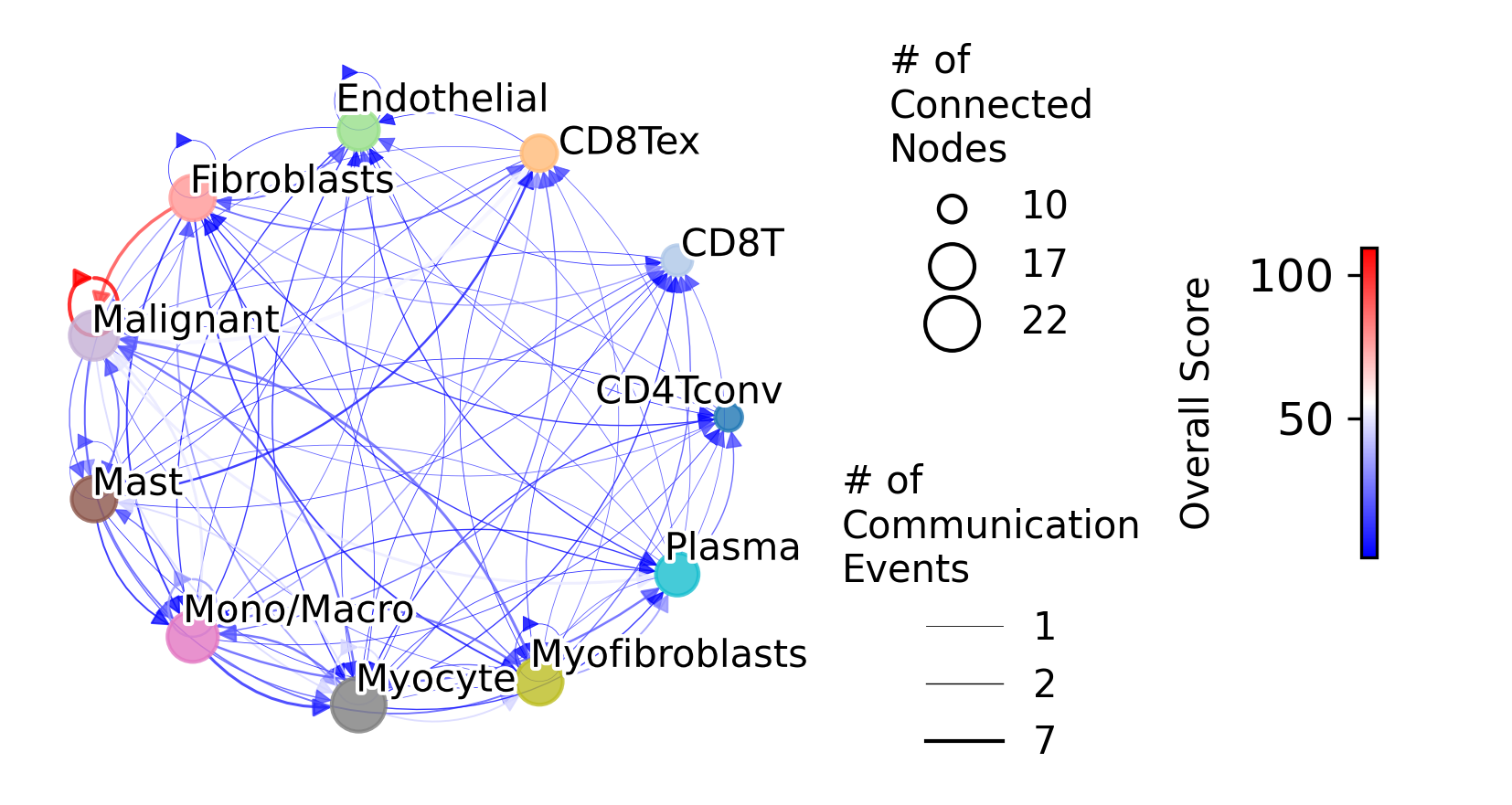
通讯网络图(Cell-cell 关系)
mebo_obj.commu_network_plot(
sender_focus=[],
metabolite_focus=[],
sensor_focus=[],
receiver_focus=[],
and_or='and',
pval_method='permutation_test_fdr',
pval_cutoff=0.05,
node_cmap='tab20',
figsize='auto',
line_cmap='bwr',
line_color_vmin=None,
line_color_vmax=None,
linewidth_norm=(0.2, 1),
linewidth_value_range=None,
node_size_norm=(50, 200),
node_value_range=None,
adjust_text_pos_node=True,
node_text_hidden=False,
node_text_font=10,
save='commu_network.pdf',
show_plot=True,
comm_score_col='Commu_Score',
comm_score_cutoff=0,
text_outline=True,
return_fig=False
)
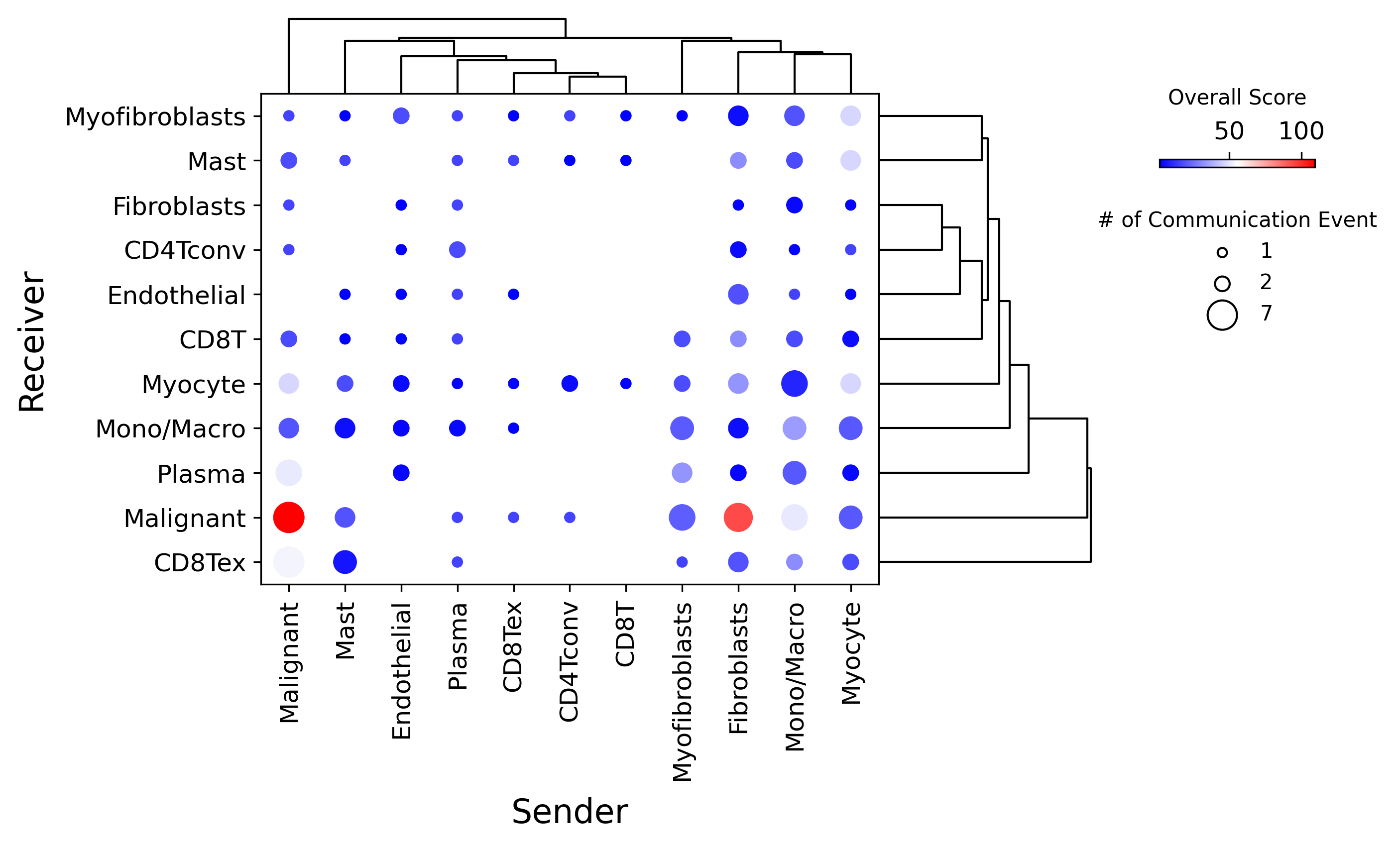
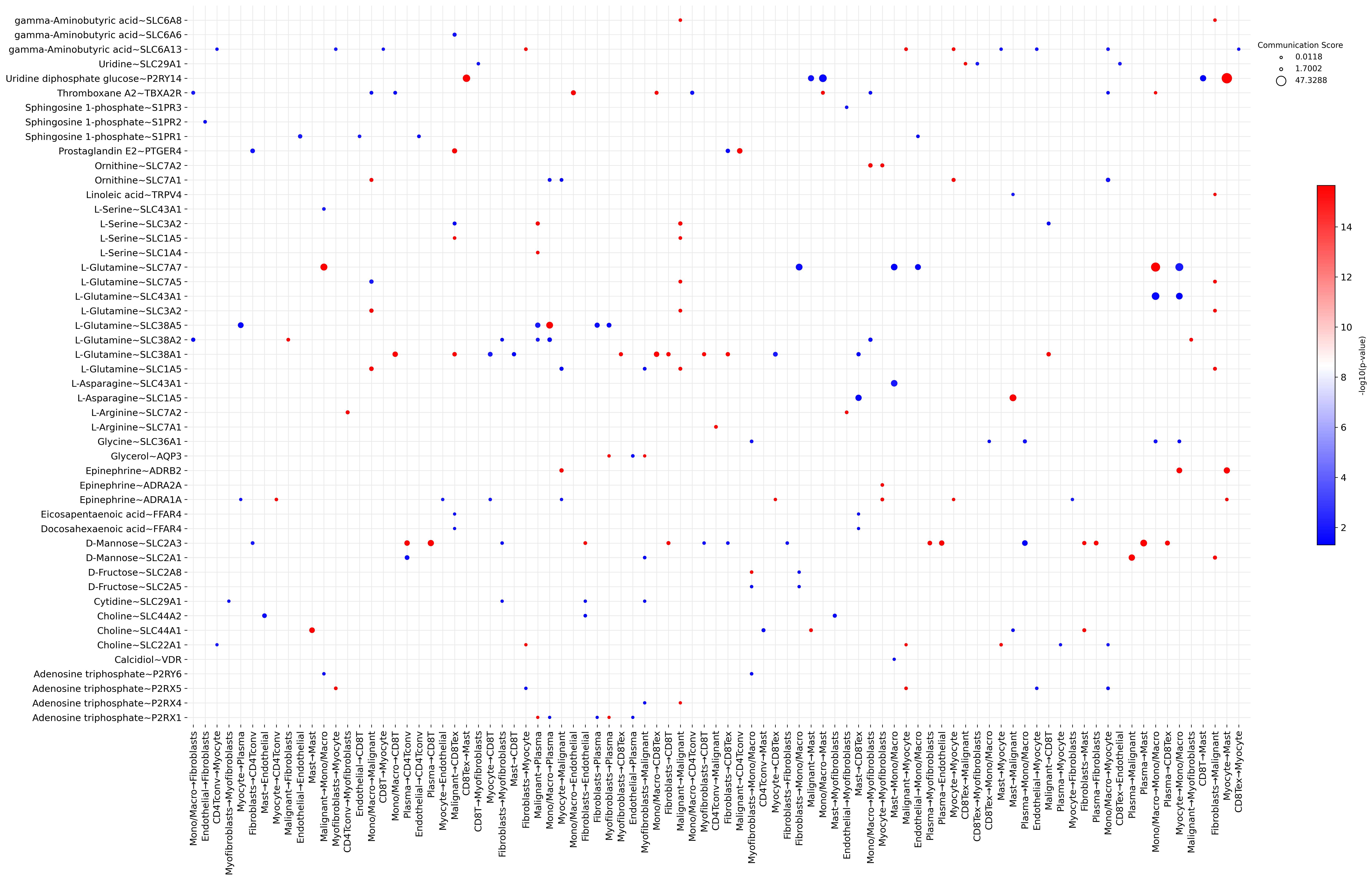
Dot 图(细胞对 vs 通讯数量)
mebo_obj.count_dot_plot(
pval_method='permutation_test_fdr',
pval_cutoff=0.05,
cmap='bwr',
figsize='auto',
save='count_dotplot.pdf',
dot_size_norm=(20, 200),
dot_value_range=None,
dot_color_vmin=None,
dot_color_vmax=None,
show_plot=True,
comm_score_col='Commu_Score',
comm_score_cutoff=0,
dendrogram_cluster=True,
sender_order=[],
receiver_order=[],
return_fig=False
)
Dot 图(细节化)
mebo_obj.commu_dotmap(
sender_focus=[],
metabolite_focus=[],
sensor_focus=[],
receiver_focus=[],
and_or='and',
pval_method='permutation_test_fdr',
pval_cutoff=0.05,
figsize='auto',
cmap='bwr',
cmap_vmin = None,
cmap_vmax = None,
cellpair_order=[],
met_sensor_order=[],
dot_size_norm=(10, 150),
save=None,
show_plot=True,
comm_score_col='Commu_Score',
comm_score_range = None,
comm_score_cutoff=0,
swap_axis = False,
return_fig = False
)
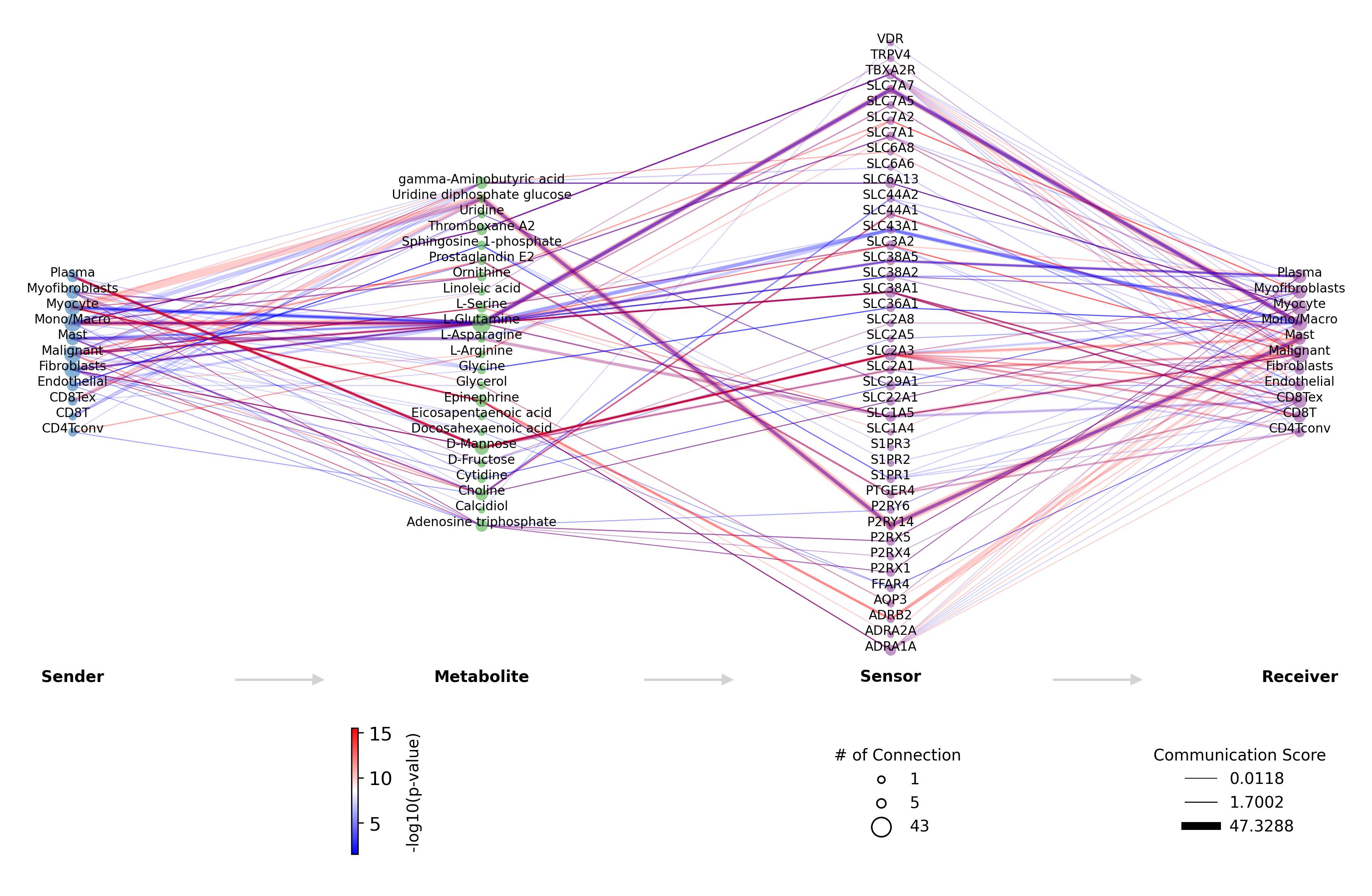
流图(Sender ➝ Metabolite ➝ Sensor ➝ Receiver)
mebo_obj.FlowPlot(
pval_method='permutation_test_fdr',
pval_cutoff=0.05,
sender_focus=[],
metabolite_focus=[],
sensor_focus=[],
receiver_focus=[],
remove_unrelevant=False,
and_or='and',
node_label_size=8,
node_alpha=0.6,
figsize='auto',
node_cmap='Set1',
line_cmap='bwr',
line_cmap_vmin=None,
line_cmap_vmax=15.5,
node_size_norm=(20, 150),
node_value_range=None,
linewidth_norm=(0.5, 5),
linewidth_value_range=None,
save='flowplot.pdf',
show_plot=True,
comm_score_col='Commu_Score',
comm_score_cutoff=0,
text_outline=False,
return_fig=False
)
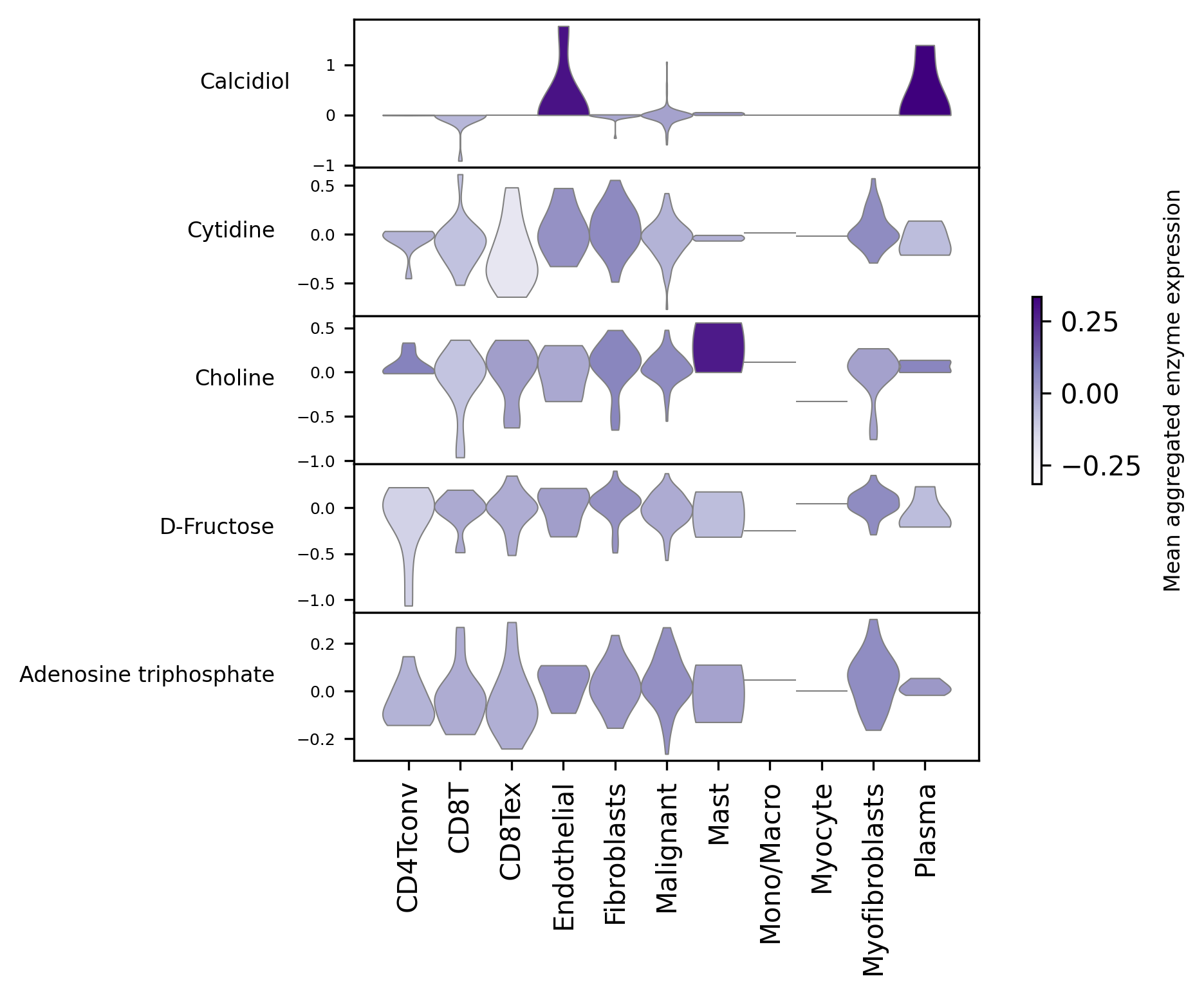
代谢物(enzyme)表达可视化
commu_df = mebo_obj.commu_res.copy()
good_met = commu_df[commu_df['permutation_test_fdr'] <= 0.05]['Metabolite_Name'].sort_values().unique()
mebo_obj.violin_plot(
sensor_or_met=good_met[:5], # 前五个显著代谢物
cell_focus=[],
cell_order=[],
row_zscore=False,
cmap=None,
vmin=None,
vmax=None,
figsize='auto',
cbar_title='',
save='violin_metabolites.pdf',
show_plot=True
)
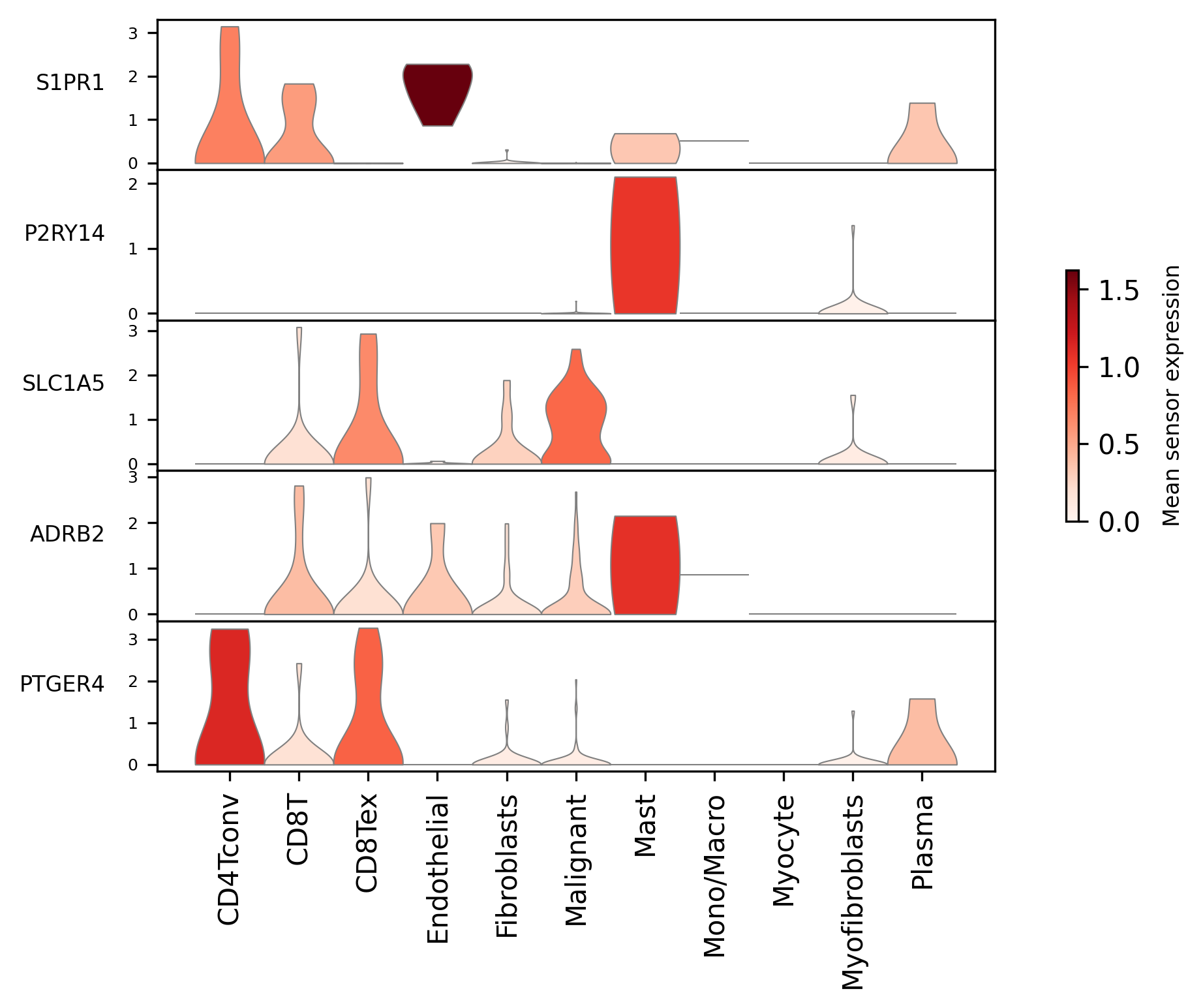
传感器(sensor)表达可视化
good_sensor = commu_df[(commu_df['permutation_test_fdr']<=0.05)]['Sensor'].sort_values().unique()
mebo_obj.violin_plot(
sensor_or_met=good_sensor[:5],## only top 5 as example
cell_focus=[],
cell_order = [],
row_zscore = False,
cmap=None,
vmin=None,
vmax=None,
figsize='auto',
cbar_title='',
save=None,
show_plot=True
)
还有第七部份是结果导出与图像保存,还有一些修改参数、过滤通讯、重新约束分析的代码大家可以参考一下原文。
这篇是非常民间的代码,突破传统配体-受体模型的局限,在单细胞分辨率下系统解析代谢物驱动的细胞通讯网络。其灵活的输入格式、完善的可视化接口与可拓展的代谢流整合功能,使其不仅适用于基础研究,也可广泛应用于肿瘤免疫、代谢疾病及再生医学等多种生物医学场景。
更多推荐
 已为社区贡献4条内容
已为社区贡献4条内容







所有评论(0)